Methoden
Datenerfassung
ARS basiert auf den Ergebnissen der Erregeridentifizierung und Resistenztestung im Rahmen der Routinediagnostik der teilnehmenden Laboratorien. Die Datenerfassung und Dokumentation findet im mikrobiologischen Labor und teilweise bereits zuvor bei der Einsendung von Proben an das Labor statt.
Variablenplan
Basis für die Übermittlung von Daten vom Labor an das RKI ist der ARS-Variablenplan. Er umfasst neben den Ergebnissen der mikrobiologischen Diagnostik Angaben zur Probe, zum Einsender der Probe und zur Patientin/zum Patienten.
Die folgende Abbildung gibt einen vereinfachten schematischen Überblick über die Datenstruktur:
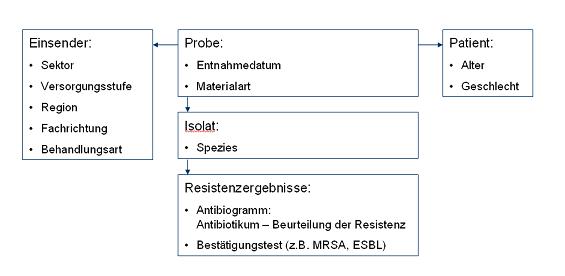 Schematischer Überblick über die Datenstruktur
Quelle: © Robert Koch-Institut
Schematischer Überblick über die Datenstruktur
Quelle: © Robert Koch-Institut
Limitationen
Die Organisation von ARS als laborgestützte Surveillance hat Limitationen bei der Datenerhebung zur Folge. Daten, die üblicherweise nicht im Labor verfügbar sind, können nicht berücksichtigt werden.
Dies betrifft insbesondere klinische Informationen zur Patientin/zum Patienten, von der/dem die Proben entnommen wurden wie Angaben zu Diagnosen, Antibiotikatherapie und Aufnahmedatum bei stationären PatientInnen.
Damit ist es im Rahmen von ARS nicht möglich, zwischen Infektionen und Kolonisationen mit einem Erreger zu unterscheiden; ebenso ist eine Trennung von nosokomialen und außerhalb des Gesundheitssystems erworbenen Infektionen (community acquired infections) nicht möglich.
Datentransfer-Datenschutz
Der Variablenplan wurde zu einer sog. XML-Schema-Definition formalisiert, die den relevanten Datenausschnitt aus einem Quellformat - dem Labor-Informations-System (LIS) - in ein standardisiertes Zielformat abbildet.
Für die am weitesten verbreitete Labor-Statistik-Software ist ein Exportmodul entwickelt worden, das nach einer einmaligen laborspezifischen Konfiguration den Datentransfer vom Labor zum RKI automatisiert.
Für andere Softwareprodukte kann zur Entwicklung von Schnittstellen zum Datentransfer die technische Dokumentation angefordert werden (ARS@rki.de).
Die Übermittlung der Labordaten an das RKI erfolgt auf Basis von § 13 Absatz 4 Infektionsschutzgesetz.
Für die epidemiologische Auswertung ist eine Zuordnung der untersuchten Proben zur Patientin/zum Patienten, von der/dem die Probe entnommen wurde, erforderlich,
um einerseits Mehrfachuntersuchungen statistisch kontrollieren zu können und andererseits Zusammenhänge zwischen Patientenmerkmalen und Resistenzen analysieren zu können.
Das RKI erhält die Angaben Geburtsmonat, Geburtsjahr und Geschlecht, die einer pseudonymisierten Patienten-ID zuzuordnen sind.
Diese pseudonymisierte Patienten-ID setzt sich aus einer eindeutigen ID des Datensenders und einer fortlaufenden Sequenz innerhalb der Datenbank des Datensenders zusammen.
Dadurch ist eine Zusammenführung von Person und Daten nur innerhalb der Datenbank des Datensenders möglich, nicht aber für das Robert Koch-Institut.
Angaben zu Gesundheitseinrichtungen, die Proben einsenden – Landkreis des Standorts sowie Versorgungsstufe bei Krankenhäusern bzw. Fachrichtung bei Arztpraxen - sind ebenfalls pseudonymisiert.
Zur Wahrung der Vertraulichkeit wird in allen Auswertungen so aggregiert, dass eine Identifizierung von einzelnen Einrichtungen ausgeschlossen ist und niemals die Daten einer einzelnen Einrichtung als Ergebnis einer Datenabfrage ausgegeben werden.
Datenvalidierung
Die Rohdaten, die von den teilnehmenden Laboren überwiegend täglich, z.T. auch wöchentlich übermittelt werden, werden in einem Data Warehouse gespeichert und weiterverarbeitet.
Als zentrales Instrument der Qualitätssicherung wird einmal jährlich eine Prüfung der Daten auf Vollständigkeit und Plausibilität vorgenommen.
Dazu erhalten die teilnehmenden Labore ein strukturiertes Feedback zu den von ihnen für das jeweils abgelaufene Jahr übermittelten Daten zur Prüfung.
Der Prozess endet mit der Freigabe oder Sperrung der Daten durch das Labor; nur vom Labor freigegebenen Daten werden in die ARS-Datenbank übernommen,
die die Basis für die Standardauswertungen zu Erreger- und Resistenzhäufigkeiten bildet.
Exkurs: Mikrobiologische Diagnostik
Als Teilnahmevoraussetzung gilt die Einhaltung der Mikrobiologisch-Infektiologischen Qualitätsstandards (MIQ) der
Deutschen Gesellschaft für Hygiene und Mikrobiologie (DGHM).
Alle an ARS beteiligten Labore erfüllen im Rahmen der Akkreditierung und Zertifizierung Kriterien der internen und externen Qualitätssicherung.
Innerhalb des von den MIQ vorgegebenen Rahmens gibt es eine Vielzahl von Methoden, die zum Einsatz kommen.
Für die Surveillance stellt die Heterogenität der Methoden und Normen im Hinblick auf die Vergleichbarkeit von Testergebnissen aus verschiedenen Laboratorien eine Herausforderung dar.
International existieren verschiedene Standards für die Durchführung und Bewertung der Ergebnisse von Empfindlichkeitsprüfungen:
In Deutschland war lange Jahre die Norm 58940 "Empfindlichkeitsprüfung von mikrobiellen Krankheitserregern gegen Chemotherapeutika" des Normenausschusses Medizin im DIN (NAMed) weit verbreitet;
ebenso die Standards des US-amerikanischen Clinical Laboratory Standards Institute (CLSI).
1987 wurde das European Committee on Antimicrobial Susceptibility Testing (EUCAST) mit dem Ziel der Harmonisierung der Standards in Europa gegründet.
Es bestimmt und aktualisiert Grenzwerte für die Empfindlichkeitstestung von antimikrobiellen Substanzen bei Bakterien und Pilzen zum Zweck des therapeutischen Einsatzes in der Humanmedizin in Europa sowie epidemiologische cut-offs,
die den Wildtyp eines Erregers von Isolaten mit erworbenen Resistenzmechanismen trennen, und publiziert Expertenregeln, die eine einheitliche Bewertung von Resistenzergebnissen unterstützen.
Seit 2012 fördert und unterstützt das Nationales Antibiotika-Sensitivitätstest-Komitee (NAK) die Implementierung von EUCAST in Deutschland.
In den Anfangsjahren von ARS testeten und bewerteten alle teilnehmenden Labore nach CLSI. Seit 2010 ist eine sukzessive Umstellung auf EUCAST zu beobachten. 2023 haben erstmals alle ARS-Labore nach EUCAST-Standards gearbeitet.
2018 hat das EUCAST eine Modifikation der Definition der Kategorien zur Empfindlichkeitsbewertung vorgenommen, die die Dosierung in den Mittelpunkt rückt und am 1.1.2019 in Kraft getreten ist.
Die zuvor allgemein gültigen Interpretationen der Bewertungen SIR – sensibel, intermediär sensibel und resistent – werden präzisiert und bedeuten nunmehr:
S – sensibel bei Standarddosierung (hohe Wahrscheinlichkeit eines therapeutischen Erfolges bei Verwendung einer Standarddosierung und –applikation)
I - sensibel bei erhöhter Exposition (durch erhöhte Dosierung bzw. Konzentration am Infektionsort)
R – resistent (hohe Wahrscheinlichkeit des Therapieversagens auch bei erhöhter Exposition).
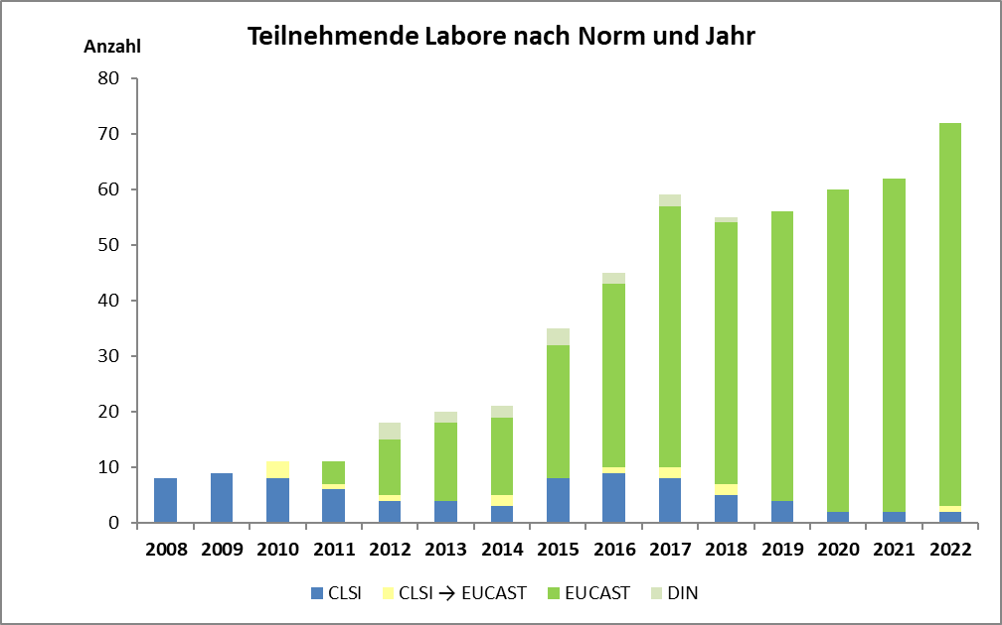 Anzahl Labore nach Jahr und Norm
Quelle: © Robert Koch-Institut
Anzahl Labore nach Jahr und Norm
Quelle: © Robert Koch-Institut
nach oben